15. Epidemics on temporal networks#
The temporal dynamics of networks affect dynamical processes defined one them
In 2001, Liljeros and collaborators found that the network of human sexual contacts is “scale-free” concluding that STI can be hard to eradicate because of such property.
See: https://www.nature.com/nature/journal/v411/n6840/full/411907a0.html
However, their conclusions were only partially true because the network of human sexual contacts is a dynamical network and temporal fluctuations are very important when considering a dynamical processes that take place on top of it.
In 2011, Rocha and collaborators published a temporal dataset of sexual contacts in Brazil: http://journals.plos.org/ploscompbiol/article?id=10.1371/journal.pcbi.1001109. We use this dataset to simulate an epidemic process (SI) and compare results between the static aggregated network and the dynamic network.
import networkx as nx
from collections import defaultdict
# %#pylab inline
%pylab is deprecated, use %matplotlib inline and import the required libraries.
Populating the interactive namespace from numpy and matplotlib
import seaborn as sns
filepath = "./../network_data/Dataset_sexual_network.csv"
edgelist = defaultdict(list)
finput = open(filepath, "r")
for line in finput.readlines():
if line[0] != "#": # not a comment
s = line.strip().split(";")
day = int(s[2])
edge = (int(s[0]), int(s[1]))
edgelist[day].append(edge)
finput.close()
We first create the aggregated network
G_agg = nx.Graph()
G_agg.disease_status = {}
daystart = 800 # we skip the transient
dayend = 1800
for d in edgelist:
if d >= daystart and d <= dayend:
links = edgelist[d]
G_agg.add_edges_from(links) # we add the link to the graph
print("The aggregated network has", len(G_agg.nodes()), "nodes")
print("The aggregated network has", len(G_agg.edges()), "links")
The aggregated network has 11306 nodes
The aggregated network has 23345 links
nx.is_connected(G_agg)
False
nx.number_connected_components(G_agg)
337
for i in nx.connected_components(G_agg):
print(len(i))
10566
3
2
2
2
2
2
2
2
3
2
4
2
2
3
2
3
2
3
2
3
2
2
2
2
2
2
2
2
3
2
2
2
2
2
2
2
2
2
2
2
2
2
3
4
2
2
6
2
4
2
4
2
3
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
3
2
2
2
2
2
2
2
3
2
2
2
2
3
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
3
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
3
2
2
2
3
2
2
2
2
2
3
2
2
2
3
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
5
2
2
3
2
2
2
2
2
3
2
2
2
8
2
5
2
3
3
2
2
2
2
4
2
2
2
2
2
3
2
2
2
2
2
2
2
2
2
2
2
3
2
2
3
2
2
2
2
2
2
2
2
2
5
3
2
2
3
2
2
2
2
2
2
2
2
2
3
2
2
2
2
2
2
3
2
2
2
2
2
2
2
2
2
3
2
3
2
2
2
2
2
2
2
2
2
2
3
2
2
2
2
2
2
2
2
2
2
3
2
2
2
2
2
2
2
2
4
2
2
2
2
2
4
2
2
2
2
2
3
2
2
2
2
2
2
3
2
2
2
2
2
2
3
2
2
2
2
2
2
2
2
2
2
2
2
2
2
2
15.1. Simulations on the time-aggregated graph#
# let's choose a random seed
import random
seed_links = edgelist[daystart]
print(len(seed_links))
random.shuffle(seed_links)
seed = seed_links[0][0]
print("The degree of the seed is", G_agg.degree(seed))
24
The degree of the seed is 2
infected_nodes = []
infected_nodes.append(seed)
for n in G_agg.nodes():
if n in infected_nodes:
G_agg.disease_status[n] = 1
# infected
else:
G_agg.disease_status[n] = 0
# susceptible
from tqdm import tqdm
Itime_agg = []
# there are 1000 days of network activity but the network does not change!
for t in tqdm(list(range(0, 1000))):
for i in infected_nodes:
for j in G_agg.neighbors(i):
if G_agg.disease_status[j] == 0:
G_agg.disease_status[j] = 1 # the probability of infection is 1!!
# ciclo per aggiornare l'elenco dei nodi infetti
infected_nodes = []
for n in G_agg.nodes():
if G_agg.disease_status[n] == 1:
infected_nodes.append(n)
# for-loop aggionrmento nodi guariti
Itime_agg.append(len(infected_nodes))
print(
"The final size of the epidemic is", float(len(infected_nodes)) / len(G_agg.nodes())
)
0%| | 0/1000 [00:00<?, ?it/s]
2%|██▊ | 17/1000 [00:00<00:05, 166.44it/s]
3%|█████▌ | 34/1000 [00:00<00:05, 161.83it/s]
5%|████████▎ | 51/1000 [00:00<00:05, 162.19it/s]
7%|███████████▏ | 68/1000 [00:00<00:05, 162.77it/s]
8%|█████████████▉ | 85/1000 [00:00<00:05, 163.02it/s]
10%|████████████████▋ | 102/1000 [00:00<00:05, 161.50it/s]
12%|███████████████████▍ | 119/1000 [00:00<00:05, 159.84it/s]
14%|██████████████████████▏ | 136/1000 [00:00<00:05, 160.93it/s]
15%|████████████████████████▉ | 153/1000 [00:00<00:05, 161.34it/s]
17%|███████████████████████████▋ | 170/1000 [00:01<00:05, 156.14it/s]
19%|██████████████████████████████▎ | 186/1000 [00:01<00:05, 151.57it/s]
20%|████████████████████████████████▉ | 202/1000 [00:01<00:05, 145.94it/s]
22%|███████████████████████████████████▎ | 217/1000 [00:01<00:05, 143.46it/s]
23%|█████████████████████████████████████▊ | 232/1000 [00:01<00:05, 143.50it/s]
25%|████████████████████████████████████████▍ | 248/1000 [00:01<00:05, 146.47it/s]
26%|██████████████████████████████████████████▊ | 263/1000 [00:01<00:05, 145.26it/s]
28%|█████████████████████████████████████████████▎ | 278/1000 [00:01<00:05, 143.07it/s]
29%|███████████████████████████████████████████████▊ | 293/1000 [00:01<00:04, 142.96it/s]
31%|██████████████████████████████████████████████████▎ | 309/1000 [00:02<00:04, 146.72it/s]
33%|█████████████████████████████████████████████████████▏ | 326/1000 [00:02<00:04, 150.99it/s]
34%|███████████████████████████████████████████████████████▋ | 342/1000 [00:02<00:04, 153.18it/s]
36%|██████████████████████████████████████████████████████████▎ | 358/1000 [00:02<00:04, 154.51it/s]
37%|████████████████████████████████████████████████████████████▉ | 374/1000 [00:02<00:04, 152.73it/s]
39%|███████████████████████████████████████████████████████████████▌ | 390/1000 [00:02<00:04, 148.76it/s]
40%|██████████████████████████████████████████████████████████████████ | 405/1000 [00:02<00:04, 146.95it/s]
42%|████████████████████████████████████████████████████████████████████▍ | 420/1000 [00:02<00:03, 145.74it/s]
44%|██████████████████████████████████████████████████████████████████████▉ | 435/1000 [00:02<00:03, 146.14it/s]
45%|█████████████████████████████████████████████████████████████████████████▌ | 451/1000 [00:02<00:03, 148.88it/s]
47%|████████████████████████████████████████████████████████████████████████████ | 467/1000 [00:03<00:03, 149.76it/s]
48%|██████████████████████████████████████████████████████████████████████████████▌ | 482/1000 [00:03<00:03, 147.51it/s]
50%|█████████████████████████████████████████████████████████████████████████████████ | 497/1000 [00:03<00:03, 145.53it/s]
51%|███████████████████████████████████████████████████████████████████████████████████▍ | 512/1000 [00:03<00:03, 143.27it/s]
53%|█████████████████████████████████████████████████████████████████████████████████████▉ | 527/1000 [00:03<00:03, 142.69it/s]
54%|████████████████████████████████████████████████████████████████████████████████████████▌ | 543/1000 [00:03<00:03, 145.52it/s]
56%|███████████████████████████████████████████████████████████████████████████████████████████ | 559/1000 [00:03<00:02, 147.80it/s]
57%|█████████████████████████████████████████████████████████████████████████████████████████████▋ | 575/1000 [00:03<00:02, 150.05it/s]
59%|████████████████████████████████████████████████████████████████████████████████████████████████▎ | 591/1000 [00:03<00:02, 152.72it/s]
61%|███████████████████████████████████████████████████████████████████████████████████████████████████ | 608/1000 [00:04<00:02, 156.41it/s]
62%|█████████████████████████████████████████████████████████████████████████████████████████████████████▋ | 624/1000 [00:04<00:02, 156.12it/s]
64%|████████████████████████████████████████████████████████████████████████████████████████████████████████▎ | 640/1000 [00:04<00:02, 156.81it/s]
66%|██████████████████████████████████████████████████████████████████████████████████████████████████████████▉ | 656/1000 [00:04<00:02, 157.38it/s]
67%|█████████████████████████████████████████████████████████████████████████████████████████████████████████████▌ | 672/1000 [00:04<00:02, 157.79it/s]
69%|████████████████████████████████████████████████████████████████████████████████████████████████████████████████▏ | 688/1000 [00:04<00:01, 157.83it/s]
70%|██████████████████████████████████████████████████████████████████████████████████████████████████████████████████▉ | 705/1000 [00:04<00:01, 160.67it/s]
72%|█████████████████████████████████████████████████████████████████████████████████████████████████████████████████████▋ | 722/1000 [00:04<00:01, 157.76it/s]
74%|████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████▎ | 738/1000 [00:04<00:01, 152.76it/s]
75%|██████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████▉ | 754/1000 [00:04<00:01, 149.89it/s]
77%|█████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████▌ | 770/1000 [00:05<00:01, 147.87it/s]
78%|███████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████▉ | 785/1000 [00:05<00:01, 145.95it/s]
80%|██████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████▍ | 800/1000 [00:05<00:01, 144.67it/s]
82%|█████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████ | 816/1000 [00:05<00:01, 146.77it/s]
83%|███████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████▍ | 831/1000 [00:05<00:01, 147.48it/s]
85%|██████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████▏ | 848/1000 [00:05<00:01, 151.91it/s]
86%|████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████▊ | 864/1000 [00:05<00:00, 153.49it/s]
88%|███████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████▌ | 881/1000 [00:05<00:00, 156.55it/s]
90%|██████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████▎ | 898/1000 [00:05<00:00, 158.70it/s]
92%|█████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████▏ | 915/1000 [00:06<00:00, 159.96it/s]
93%|███████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████▊ | 931/1000 [00:06<00:00, 154.97it/s]
95%|██████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████▎ | 947/1000 [00:06<00:00, 150.69it/s]
96%|████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████▉ | 963/1000 [00:06<00:00, 148.67it/s]
98%|███████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████▌ | 979/1000 [00:06<00:00, 149.91it/s]
100%|██████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████| 1000/1000 [00:06<00:00, 151.69it/s]
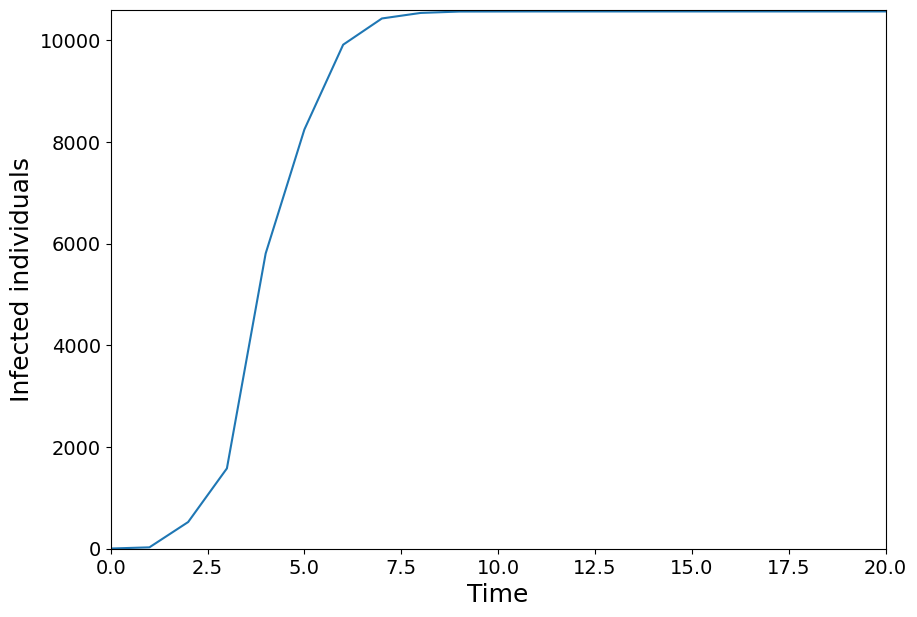
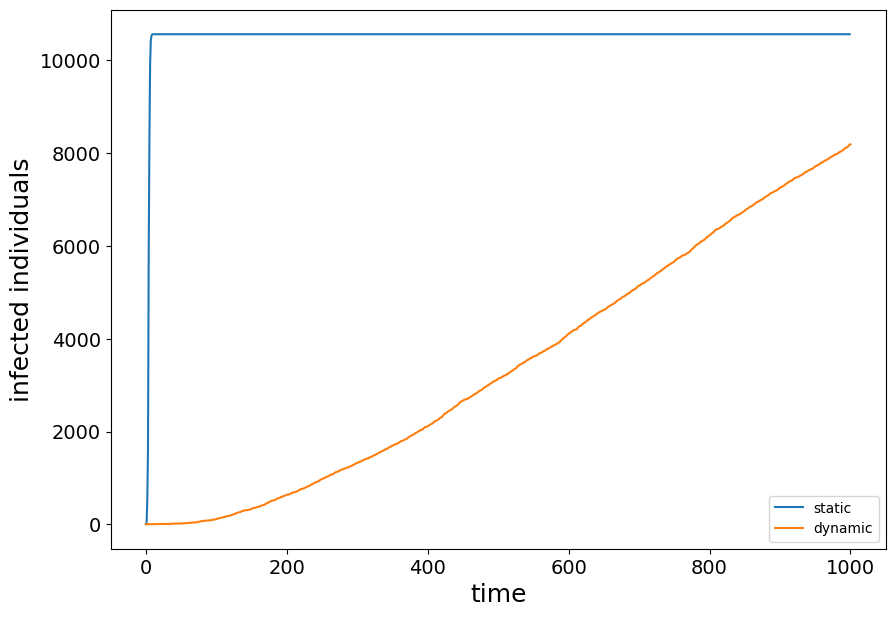
The final size of the epidemic is 0.9345480275959668
plt.figure(figsize=(10, 7))
plt.xlabel("Time", fontsize=18)
plt.ylabel("Infected individuals", fontsize=18)
plt.xticks(fontsize=14)
plt.yticks(fontsize=14)
plt.plot(range(0, len(Itime_agg)), Itime_agg)
plt.axis([0, 20, -1, 10600])
(0.0, 20.0, -1.0, 10600.0)
15.2. Simulations on the full dynamic network#
G_dyn = nx.Graph()
G_dyn.disease_status = {}
seed_links = edgelist[daystart]
random.shuffle(seed_links)
seed = seed_links[0][0]
seed
98
infected_nodes = []
infected_nodes.append(seed)
G_dyn.add_edges_from(seed_links) # we consider only the links that are active on day 0
for n in G_dyn.nodes():
if n in infected_nodes:
G_dyn.disease_status[n] = 1
# infected
else:
G_dyn.disease_status[n] = 0
# susceptible
Itime_dyn = []
for t in range(daystart, dayend + 1):
links = edgelist[t] # these are the links active on day t
if t == daystart:
print("The temporal network has", len(G_dyn.nodes()), "nodes on day", daystart)
print("The temporal network has", len(G_dyn.edges()), "edges on day", daystart)
print("The degree of the seed is", G_dyn.degree(seed))
else:
G_dyn.add_edges_from(links)
for e in links:
if e[0] not in G_dyn.disease_status:
G_dyn.disease_status[e[0]] = 0
if e[1] not in G_dyn.disease_status:
G_dyn.disease_status[e[1]] = 0
# ciclo sui nodi infetti per la trasmissione
for i in infected_nodes:
for j in G_dyn.neighbors(i):
if G_dyn.disease_status[j] == 0:
G_dyn.disease_status[j] = 1
# ciclo per aggiornare l'elenco dei nodi infetti
infected_nodes = []
for n in G_dyn.nodes():
if G_dyn.disease_status[n] == 1:
infected_nodes.append(n)
Itime_dyn.append(len(infected_nodes))
# stampo il numero di nodi infetti a ogni time-step
G_dyn.remove_edges_from(links)
print(
"The final size of the epidemic is", float(len(infected_nodes)) / len(G_dyn.nodes())
)
The temporal network has 22 nodes on day 800
The temporal network has 12 edges on day 800
The degree of the seed is 1
The final size of the epidemic is 0.7244825756235627
plt.figure(figsize=(10, 7))
plt.xticks(fontsize=14)
plt.yticks(fontsize=14)
plt.xlabel("time", fontsize=18)
plt.ylabel("infected individuals", fontsize=18)
plt.plot(range(0, len(Itime_agg)), Itime_agg, label="static")
plt.plot(range(0, len(Itime_dyn)), Itime_dyn, label="dynamic")
plt.legend()
# plt.axis([0,365,-1,10600])
<matplotlib.legend.Legend at 0x1779a3a30>
15.3. homeworks#
does it accelerate if randomized?
does it if clustering coefficient (triangles)?
does it if temporal clustering?

